Research Topics
Basic research
- Minimal community of gut microbiota
以大蠟蛾建立腸道微生物相最小功能群定植之模式動物、並以奈米孔定序分析定植微生物相之動態 (國科會新進人員研究計畫)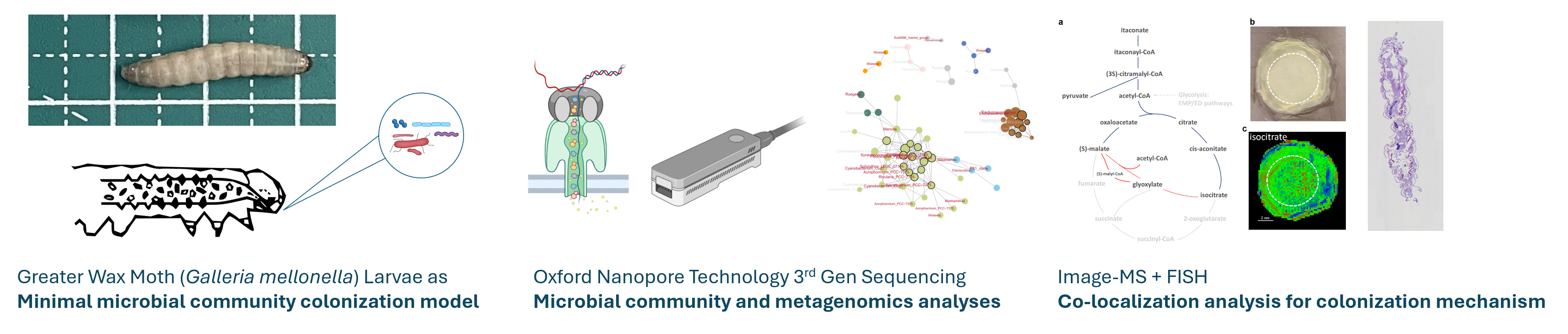
Clinical microbiome
- Metabolic-associated fatty live disease 代謝異常脂肪肝 (MAFLD)
Gut microbiota of lean MAFLD patients
Bioinformatics for microbiome analysis
- Kmer taxonomic unit (KTU) algorithm development
- A Novel Approach to Differential Expression Analysis of Co-occurrence Networks for Small-sampled microbiome Data
- Cross-domain metagenome binning algorithm development
- Marine metagenome analysis: Metagenomic Survey for North-Western Pacific Ocean using 2nd/3rd-Generation Hybrid Sequencing
collaborate with Dr. Sen-Ling Tan (Academia Sinica).
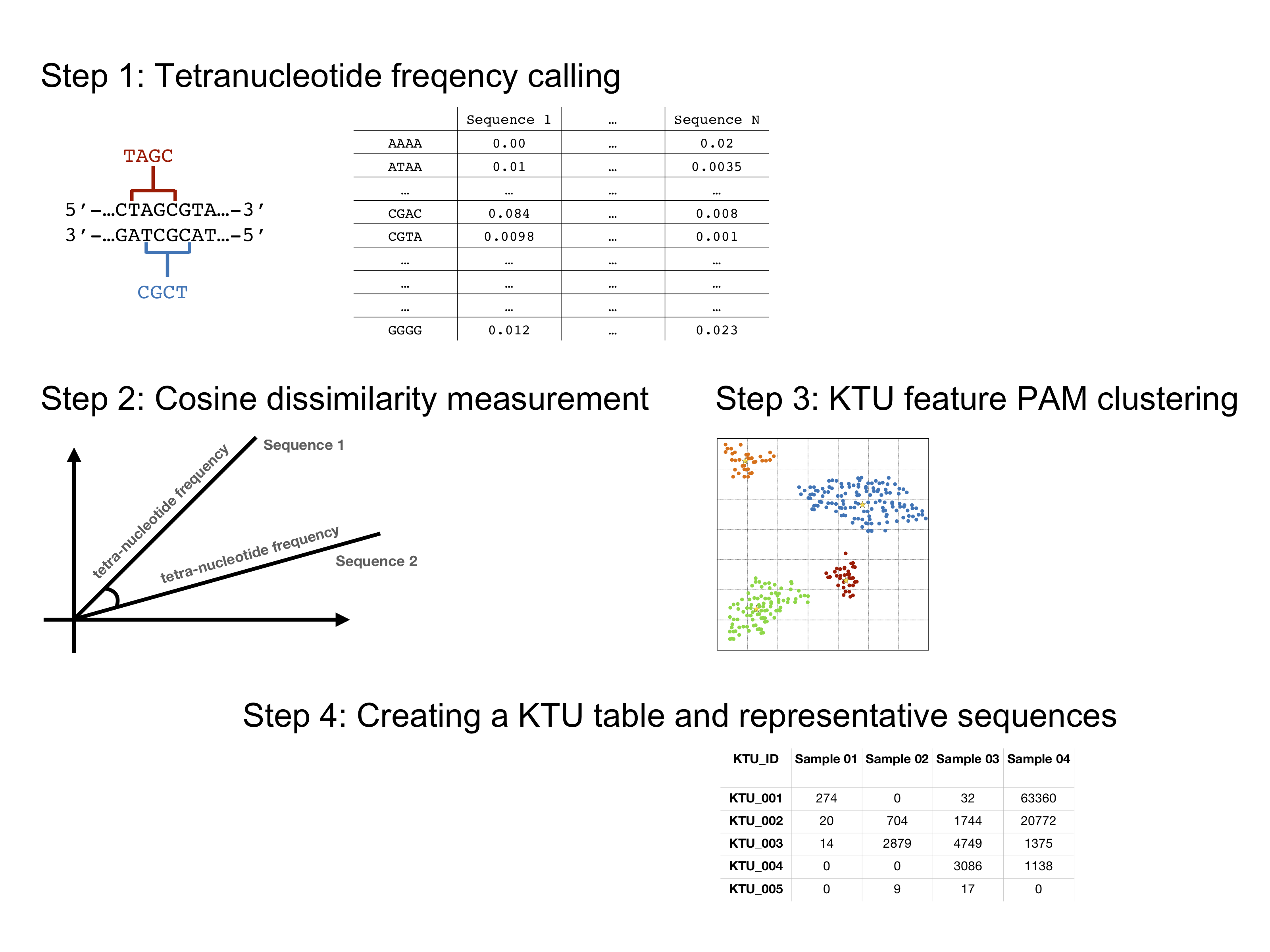

近年來,16S amplicon 的microbiome研究分析工具,從原本的OTU clustering 改為amplicon sequence variants (ASVs) denoising方法
,大大提升了序列辨識的解析度(單一核苷酸的解析度),堪稱能鑑別菌株(strains)。不過這樣的高解析度,對微生物生態學家卻產生很大的困擾,
單一核苷酸的解析度忽略了物種本有的「種內變異」,並且產生太多的0,因此使用ASVs來分析微生物相與環境變數的相關性時,相關性不僅下降了,甚至變成沒有相關;
此外還可能高估了樣本的alpha 多樣性。此外,即使16S rRNA基因的演化速率穩定,但並非每種物種的演化速度都等速(所以單用一個序列相似度閾值的OTU clustering也會被詬病)。
我們開發了KTU,是基於k-mer將ASVs再重新類聚(reclustering),並不是完全摒棄ASVs的優點,反而是藉助ASV/denoising的高解析度與錯誤校正,然後根據k-mer,
也就是genome signature或是每種物種的codon usage bias的特徵,再由機器學習的類聚方法K-medoids (Partitioning Around Medoids (PAM))找尋最佳的物種數。
那效果如何呢?
我們用一篇酸種麵團菌相的data來驗證KTU的功效。酸種麵團的酸味來源來自於多種乳酸桿菌(Lactobacillus),不過Lactobacillus這個分類在2020年有很大的革新,被拆成25個新的屬(genus/genera),
而且乳酸桿菌有很多次分群種,每個次分群中的16S rRNA gene相似度都高達99%以上。利用KTU我們不但可以將次分群內的屬、種幾近完美地重新類聚在一起,
也不影響整個data的親緣距離多樣性(Faith's Phylogenetic diversity)。
KTU R package的GitHub連結:https://github.com/poyuliu/KTU/
論文全文連結:http://doi.org/10.1111/2041-210X.13758
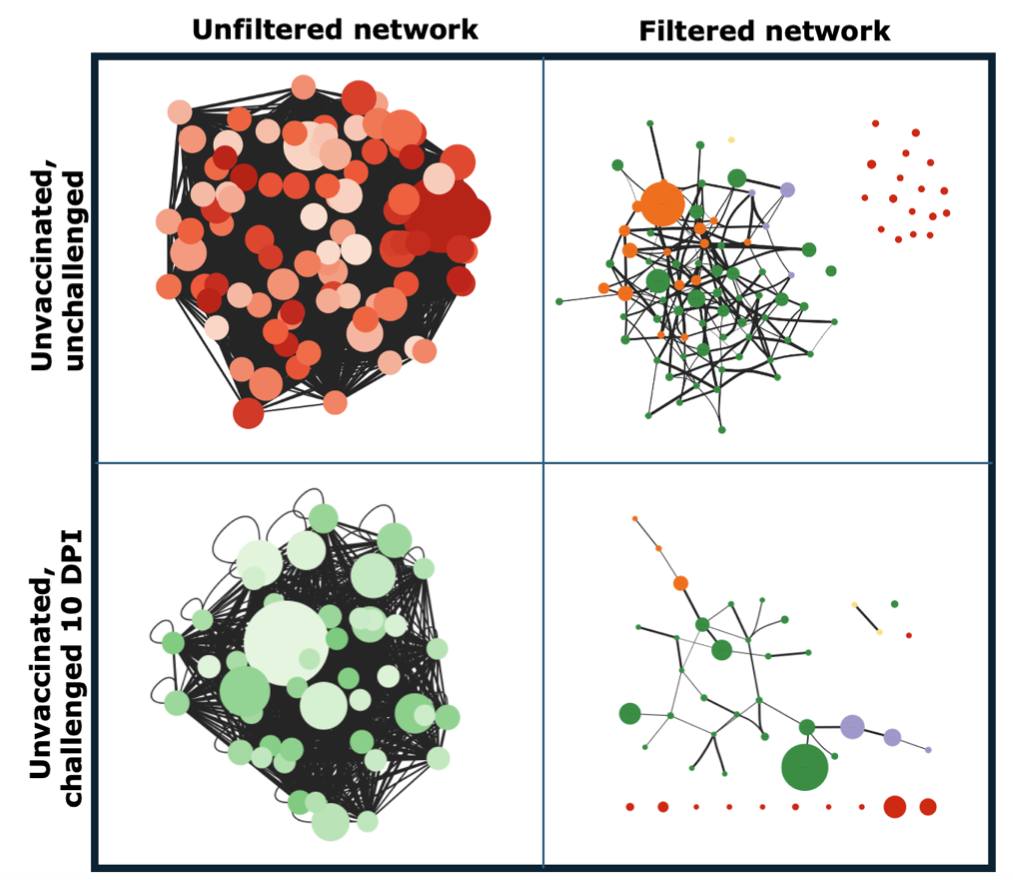
我們與英國IBM研究院(IBM Research UK)、利物浦大學及英國高性能運算中心(Hartree Centre)開發的這項微生物相網絡分析工具是針對小樣本數據中的微生物共現網路(co-occurrence networks)進行分析,
並以感染 Eimeria tenella寄生蟲的雞腸道菌相作為研究對象。
我們透過蒙地卡羅方法建立統計模型,有效去除虛假關聯(spurious associations);使用 bootstrapping 來解決小樣本數據集帶來的統計不穩定性問題。這方法可以精準篩選出具有生物學意義的微生物互動。
結果顯示,在不同感染階段中存在一組穩定的核心微生物群,可能在維持腸道生態平衡方面扮演關鍵角色。
此外,透過網路聚類分析,發現感染進程會顯著改變微生物群落的結構與互動模式。這項研究提供了深入了解腸道微生物如何應對感染的科學依據,並為未來疫苗研發、疾病監測以及腸道健康管理提供重要參考。
此方法亦具備廣泛應用潛力,適用於其他小樣本的微生物研究,有助於推動精準醫學與生物科技的發展。
論文全文連結:https://ieeexplore.ieee.org/abstract/document/10938913
Veterinary microbiome
- Multi-omics investigation for evaluating efficacy of anticoccidiosis vaccine
collaborate with Royal Veterinary College, University of London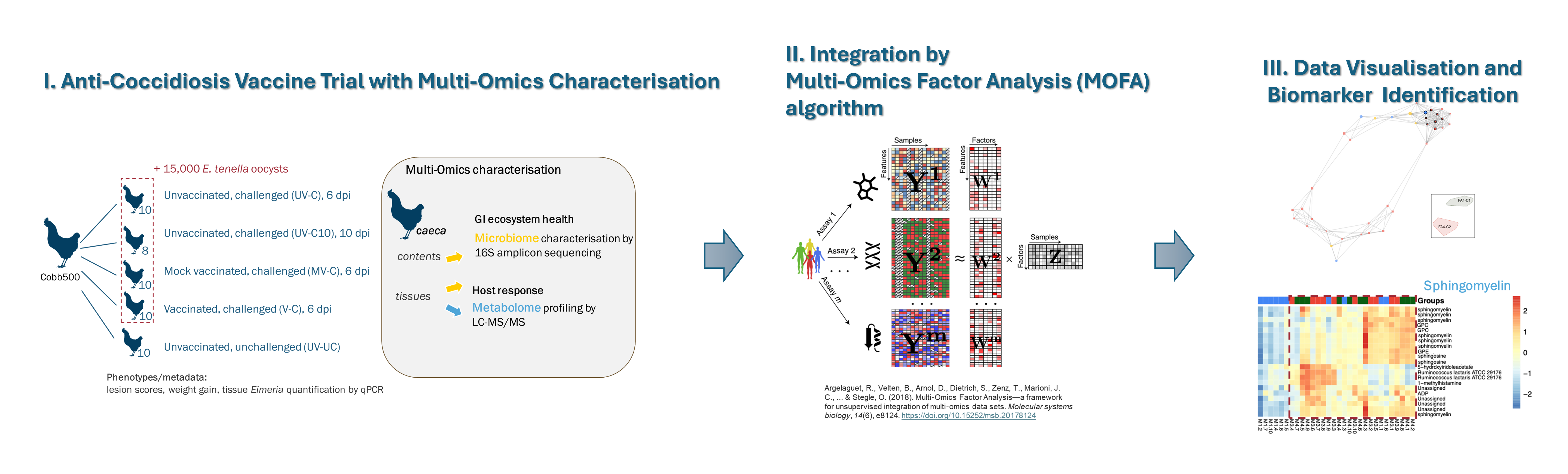
球蟲病(Coccidiosis)由艾美球蟲(Eimeria spp.)寄生蟲引起,影響家禽健康與經濟效益。本研究與英國倫敦大學皇家獸醫學院、倫敦衛生及熱帶醫學研究所合作研究利用多體學工具MOFA及網路分析,整合微生物體與代謝體, 評估接種抗寄生蟲疫苗與寄生蟲感染對雞隻盲腸代謝體與微生物群的影響,發現疫苗提升有益菌群(如,雙歧桿菌Bifidobacterium spp. 和產短鏈脂肪酸的細菌),以及神經細胞和細胞膜重要成分–鞘脂類(sphingolipids), 這些都是潛在生物標記,將有助獸醫和畜產在寄生蟲病上的預防、診斷和治療。
研究成果發表於美國微生物學會《mSystems》期刊,論文全文參閱:https://journals.asm.org/doi/full/10.1128/msystems.00947-24
- Immune-mediated hematological disease in dogs is associated with alterations of the fecal microbiota
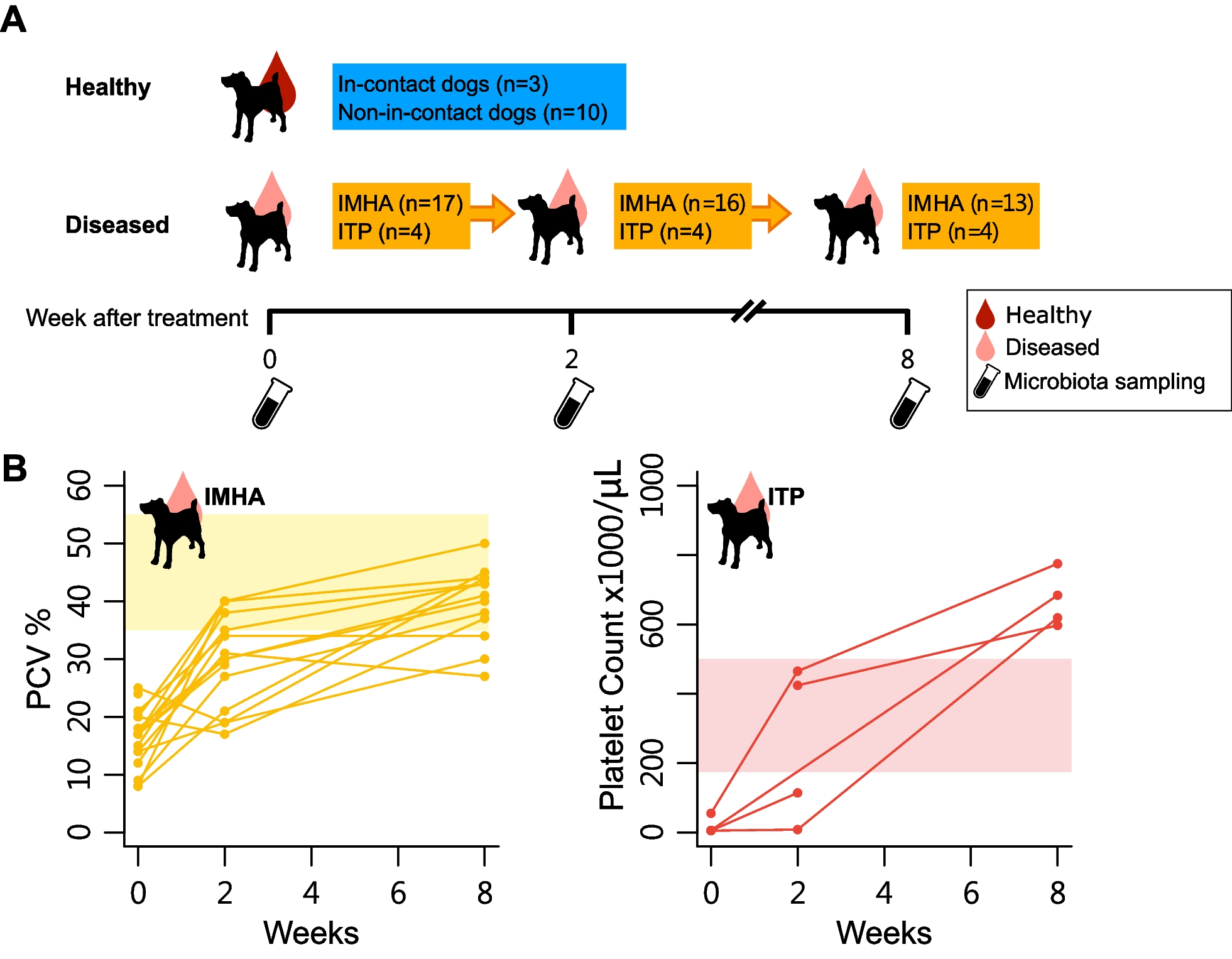
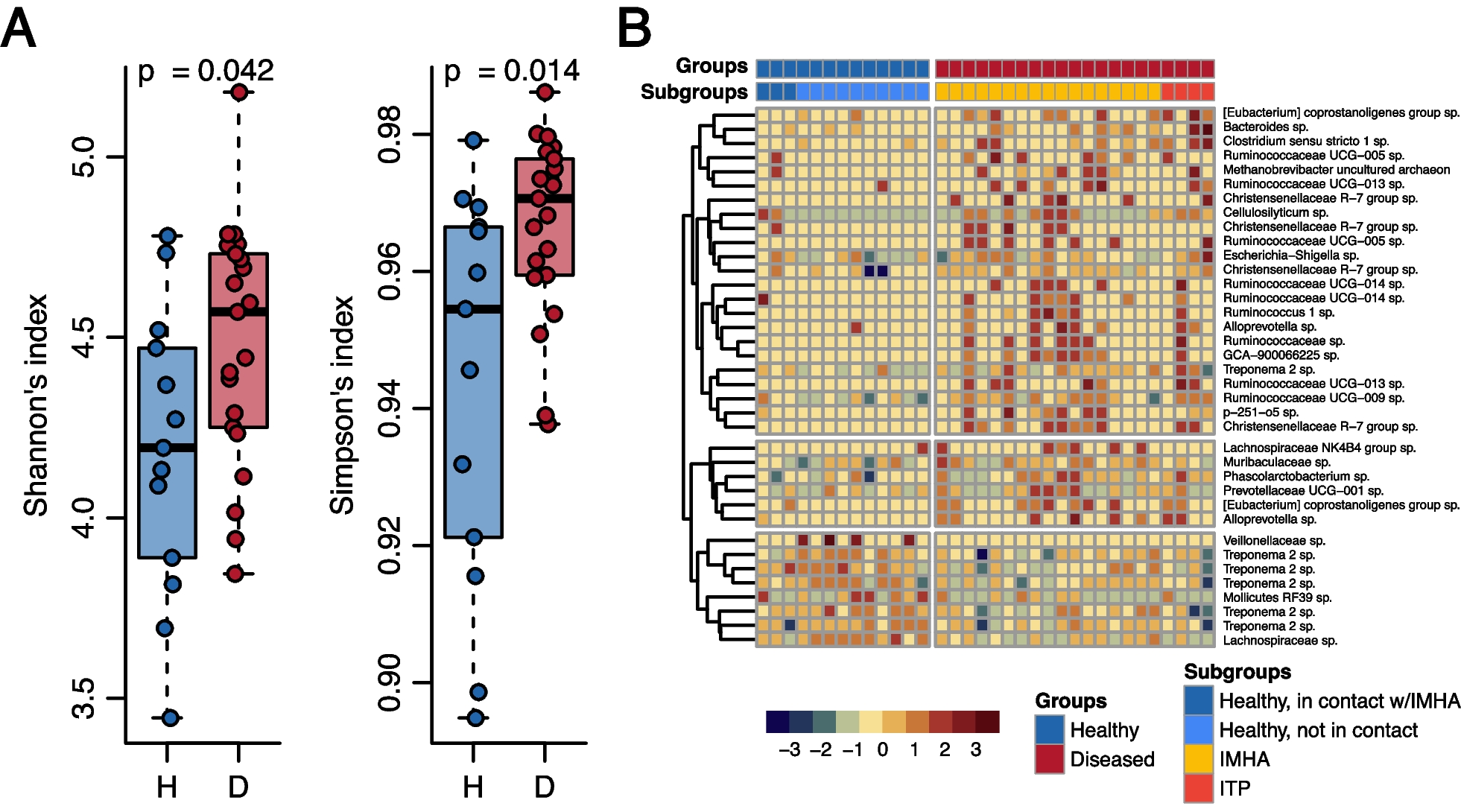
本研究與美國路易斯安那州立大學獸醫學院、賓州大學獸醫學院,以及英國倫敦大學皇家獸醫學院進行跨國合作,發現腸道微生物相與狗的自體免疫性貧血之間的複雜關係。研究成果發表於《Animal Microbiome》期刊
,並在美國獸醫血液學和輸血醫學會(Association of Veterinary Hematology and Transfusion Medicine – AVHTM)2024年第二期電子報中獲得專文報導。
本研究聚焦於狗的兩種常見自體免疫性貧血,包括溶血性貧血和血小板減少症,這兩種病症在人類自體免疫性貧血中也相當普遍,然而其確切的致病因子仍不明確。
基於此,研究團隊鎖定對宿主免疫系統發育有重要影響的腸道微生物相,探討其對自體免疫疾病發展的風險。本研究採用次世代基因定序技術,對患有這些疾病的狗在不同階段(初診以及治療後的2週和8週)
的糞便微生物相進行分析。研究納入了21隻患病狗和13隻健康對照狗,提供了自體免疫性貧血相關腸道菌相變化的全面視角。
本研究最引人注目的發現是,健康狗的微生物相中,密螺旋體屬(Treponema spp.)顯示出顯著的保護作用。
儘管同屬菌種中的梅毒螺旋體(Treponema pallidum)是梅毒的致病菌,這項研究顛覆了我們對該菌屬的傳統認知。此外,研究發現患病狗中的腐敗性梭菌(Clostridium septicum)
和潛在病原性的大腸桿菌(Escherichia coli)顯著增加,闡明了腸道微生物相失衡(生態失衡,dysbiosis)對自體免疫疾病發展的影響。此研究結果對疾病的病程提供了新興的見解和證據,
亦為診斷工具和治療策略的研究開闢了新的途徑。
論文全文參閱:https://animalmicrobiome.biomedcentral.com/articles/10.1186/s42523-023-00268-2
美國獸醫血液學和輸血醫學會專文報導:https://avhtm.org/i4a/doclibrary/getfile.cfm?doc_id=23